روسیه| توسعه یک الگوریتم تحلیل سریع برای DNA باکتریها
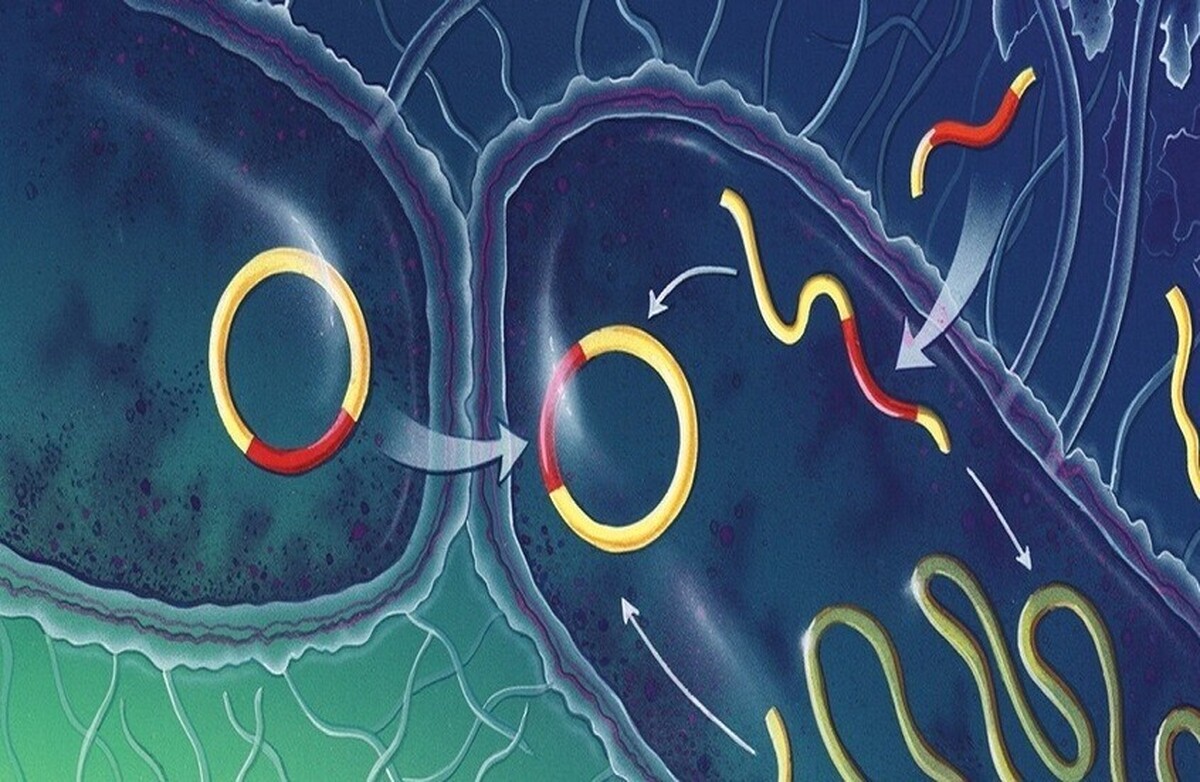
به گزارش مجله خبری نگار، دانشگاه مکانیک و میکرواپتیک از الگوریتمی به نام Strainy رونمایی کرده است که اجازه میدهد متغیرهای موجود در DNA باکتریهای درون میکروبیوم با دقت بالایی مرتب شوند و سویههای نزدیک به هم را تقسیم کنند. به لطف این توسعه، دانشمندان قادر خواهند بود سطح پاتوژنز و مقاومت آنتی بیوتیکی را در جوامع باکتریایی خاص سریعتر تعیین کنند و همچنین در مورد تکامل آنها اطلاعات بیشتری کسب کنند.
در این بیانیه یادآوری شده است که میکروبیوم مجموعهای از صدها میکروارگانیسم است که در یک محیط واحد زندگی میکنند. هر میکروارگانیسم بر انسان و محیط زیست تأثیر متفاوتی میگذارد، میتواند مضر یا مفید باشد. برای شناسایی این ویژگی ها، دانشمندان توالیهای ژنی را برای جوامع باکتریایی که به عنوان متاژنوم شناخته میشوند، "سنتز" میکنند. الگوریتمهایی که در حال حاضر مورد استفاده قرار میگیرند میتوانند این کار را انجام دهند، اما آنها فقط دادههای متوسط موجودات را منعکس میکنند و جداسازی سویههای خاص در جوامع و شناسایی ویژگیهای منحصربهفرد آنها را دشوار میکند.
دانشمندان این دانشگاه الگوریتمی را برای جمع آوری توالیهای ژن میکروبیوم در سطح نژاد ایجاد کردهاند که بیشتر جهشها در DNA میکروارگانیسمها را منعکس میکند. این الگوریتم در دو مرحله با دادههای متاژنوم تعامل دارد:
در مرحله اول، دادهها را جمع آوری میکند، قطعات DNA را با ژنوم مرجع مقایسه میکند و در صورت وجود تفاوت در یک قسمت خاص، نوع جدیدی را به توالی موجود اضافه میکند. به این ترتیب، اطلاعات مربوط به تقریبا تمام جهشهای باکتریایی در ژنوم "ثبت" میشود.
در مرحله دوم، الگوریتم با در نظر گرفتن تمام متغیرهای شناسایی شده برای ژنوم مرجع، متاژنوم اصلی را دوباره جمع میکند.
"دادههای به دست آمده میتواند نه تنها برای شناسایی سویههای موجود در میکروبیوم، بلکه برای توصیف باکتریهای موجود در آن نیز مورد استفاده قرار گیرد. به عنوان مثال، تعیین توانایی ایجاد بیماری برای موجودات دیگر و مقاومت آنتی بیوتیکی. این اطلاعات همچنین به مطالعه عمیقتر تکامل و تغییر باکتریها و همچنین یادگیری پیش بینی تغییرات احتمالی در ژنوم کمک میکند.
منبع: TASS
- لطفا از نوشتن با حروف لاتین (فینگلیش) خودداری نمایید.
- از ارسال دیدگاه های نا مرتبط با متن خبر، تکرار نظر دیگران، توهین به سایر کاربران و ارسال متن های طولانی خودداری نمایید.
- لطفا نظرات بدون بی احترامی، افترا و توهین به مسئولان، اقلیت ها، قومیت ها و ... باشد و به طور کلی مغایرتی با اصول اخلاقی و قوانین کشور نداشته باشد.
- در غیر این صورت، «نگارمگ » مطلب مورد نظر را رد یا بنا به تشخیص خود با ممیزی منتشر خواهد کرد.




- آخرین اخبار
- محبوب
- برچسب های داغ
- «آداب عروسی» میتواند به کاهش استرس و جلوگیری از احساسات منفی یا سوءتفاهمها کمک کند
- قورت دادن آدامس! چه اتفاقی برای بدن شما میافتد؟
- ویدئو| اسپانیا: چهار نفر پس از برخورد موج قدرتمند به یک استخر طبیعی کشته شدند
- ویدئو| بریژیت مکرون فعالان فمینیست را "زنان کثیف و احمق" توصیف میکند
- انتظار میرود طوفان بایرون ظرف ۲۴ ساعت خاورمیانه را درنوردد و هشدارهایی مبنی بر بارش شدید باران و جاری شدن سیل گسترده صادر شده است
- ساعات خواب «طلایی»
- هفت عادت ساده برای افزایش طول عمر و بهبود کیفیت زندگی
- مطالعه: تماشای آثار هنری اورجینال در گالریها فوراً استرس را کاهش میدهد
- مطالعه: لحظات شادی مشترک، هورمونهای استرس را در زوجها کاهش میدهد
- آیا پیادهروی در مناطق شهری میتواند همان مزایای پیادهروی در طبیعت را داشته باشد؟
- نیویورکیها بیشتر از کل کشورهای اروپایی برای سایتهای پورن هزینه میکنند
- لامنز، دروازهبان منچستریونایتد، توضیح داد که شمارههای ۸ و ۲۴ روی دستکشهایش به کوبی برایانت مربوط میشوند
- توصیهها هنگام مواجهه با خرسها: مقابله کنید، چمباتمه بزنید و آرام باشید
- سازمان ملل: تقاضا برای کولر گازی تا سال ۲۰۵۰ سه برابر خواهد شد
- محمد صلاح: به خانوادهام گفتم که برای خداحافظی با هواداران به بازی بعدی برایتون بیایند
- امیدی تازه برای مردان: یک درمان انقلابی نتایج شگفتانگیزی در بازیابی موهای از دست رفته نشان میدهد
- قلیان کشیدن خطر ابتلا به سرطان ریه را ۲ تا ۵ برابر افزایش میدهد!
- از دهان تا مغز: چگونه آرام غذا خوردن اضطراب و استرس را کاهش میدهد
- «در رقص پایانی» ... مسی و کریستیانو رونالدو کجای جام جهانی به هم خواهند رسید؟
- سورپرایز... ۷ مقصد احتمالی محمد صلاح برای ترک لیورپول
- طولانیترین خورشید گرفتگی ۲۰۲۷ را از دست ندهید!
- خرمالو ۶نفر را به اتاق عمل کشاند!
- یک نوع مصالح ساختمانی جدید که بسیار دوستدار محیط زیست است!
- رباتی انساننما که در تقلید انسان حرف ندارد!
- هوش مصنوعی فضاپیماهای ناسا را در برابر هک مقاوم کرد!
- نوسازی پوست خود را به ویتامین C بسپارید!
- غذاهای فوقفراوریشده کدامند؟ مصرف آنها عوارض دارد؟
- آزار کلامی کودک چه عواقبی دارد؟
- کاهش ۳۰ درصدی کالری دریافتی مغز را نجات میدهد!
- طراحی نانومادهای انعطافپذیر که در اثر کشش تغییر رنگ میدهد!
- ما از تمام ظرفیت مغز خود استفاده میکنیم؟
- کدام مدلهای هوش مصنوعی غیر قابل اعتمادند؟
- چگونه از شر میگرنهای آزاردهنده رها شویم؟
- موج ابتلا به آنفلوآنزا فروکش خواهد کرد؟
- کشف قدیمیترین انفجار پرتو گاما به کمک جیمز وب محقق شد!
- ثبت تشکیل امواج ترکیبی مگنون-پولارون مناسب برای رسیدن به ۶ G!
- کلید خواب شبانه در مغز شما نیست!
- خشکی و ترک خوردگی پوست در زمستان را چگونه برطرف کنیم؟
- کارتونهای تخیلی چه تاثیری بر کودکان دارند؟
- با سرعت غذا خوردن در هضم مشکل ایجاد میکند!
- استرس میتواند علت ریزش مو باشد؟
- از دست دادن حس بویایی بخاطر چه عواملی است؟
- مصرف زیاد قهوه در بزرگسالان میتواند مشکل زا شود!
- بیحوصلگی و بیانگیزگی ریشه در مغز افراد دارد!
- گاز خنده عاملی برای کاهش علائم افسردگی حاد!
- اضطراب دائمی ریشه در چه مشکلاتی دارد؟
- یک ساعت خواب ثابت کلید بهبود فشار خون!
- آلودگی هوا تاثیری بر منع شیردهی مادران دارد؟
- اشتباه هنگام استفاده از کنسروها به مرگ منجر میشود!
- مصرف قیماق قوای مادر را برمیگرداند!
- نان سالم چه ملاکهایی دارد؟
- بزرگترین چالش کنترل بیماری در ایران چیست؟
- برای رسیدن به کاهش وزن مطلوب رعایت این ۲ مورد ضروری است!
- چرا شیوه خرید و نگهداری نان اهمیت دارد؟
- بیماران مبتلا به سینوزیت برای حفظ سلامت خود در روزهای آلوده بخوانند!
- شایعترین نوع سرطان در میان زنان کدام است؟ چگونه میتوان از آن دوری کرد؟
- اسپیرولینا میتواند برای سلامتی ضرر داشته باشد؟
- نان سفید بر خلاف نانهای دیگر میتواند باعث بیماری شود!
- علائمی مانند پر ادراری را جدی بگیرید!
- مصرف اسپیرولینا میتواند به کبد شما آسیب بزند!
- اهمیت درمان به موقع بیماریهای لثه!
- چگونه بدن را در برابر سموم هوا تقویت کنیم؟
- مصرف غذاهای گرم و رطوبترسان مناسب این فصل است!
- نان کامل را چگونه نگهداری کنیم؟
- بررسی تاثیر آلودگی هوا بر سلامت پوست!
- ترکیب عسل و لیمو برای سرفه معجزه میکند!
- لباسهای جدید را قبل از پوشیدن بشویید!
- هشدار اولیه آلزایمر با بررسی این مورد!
- نخستین دستگاه پوشیدنی برای تشخیص استرس مزمن!
- چالشهای اساسی در مسیر مبارزه با سرطان کدامند؟
- مقابله با مقاومت نسبت به انسولین در دیابت نوع دوم ممکن شد!
- تغییر در عادتهای رانندگی نشانه اولیه ابتلا به بیماری آلزایمر است!
- روش خرید و نگهداری نان را بیاموزید!
- پروتکلهای بهداشتی که برای پیشگیری از آنفلوآنزا باید رعایت کنید!
- گوشی هوشمند برای بچهها خطر چاقی و افسردگی دارد!
- قلب باطری دارد؟
- طولانیترین خورشیدگرفتگی قرن بیست و یکم در این تاریخ رخ میدهد!
- نقش ویتامینهای گروه B + علائم کمبود!
- یک کشف جالب در مورد زنبورها که فاش نشده بود!
- از خواص معجزه آسای کدو حلوایی غافل نشوید!
- مارسلو الگوی خود در دنیای فوتبال را معرفی کرد
- عکس| هالند عکسی از آرشیو خود را با شیرر به اشتراک گذاشت و نوشت: «از توصیهات در سال ۲۰۲۲ متشکرم.»
- عکس| فلاویو بریاتوره در گرند پری فرمول یک با دیوید بکهام ملاقات کرد
- شانزده سال پس از مرگ او، گزارشی حقایق تکان دهندهای را در مورد کالبدشکافی این ستاره جهانی موسیقی پاپ فاش میکند
- خانواده بروس ویلیس با وخامت حال او، «تصمیمی دشوار» میگیرند
- کاردی بی از بند ناف فرزندش یک گردنبند طلا درست کرد
- مدونا مخفیانه با معشوق ۲۹ سالهاش نامزد کرده است
- کیت میدلتون و پرنس ویلیام به خاطر نحوه تشکر از کارکنانشان مورد انتقاد قرار گرفتهاند
- اشنایدر در یک نظرسنجی سریع به سوالات پاسخ داد: «کریستیانو رونالدو یا رافائل نادال؟»
- همسر سابق جنیفر آنیستون به رابطه نامشروع همسرش با یک متخصص هیپنوتیزم درمانی واکنش نشان داد
- کیران کالکین برای سومین بار پدر شد
- جزئیات رابطه عاشقانه سوفی ترنر با همسر سابق گوئینت پالترو فاش شد
- ریس ویترسپون در مورد رابطه جنیفر آنیستون با پیت: «او کسی نیست که به گذشته بچسبد.»
- رسوایی تکاندهنده درباره یک میلیاردر جهانی: او عاشق جفری اپستین، مجرم جنسی، بود، اما!
- جنیفر لارنس توضیح داد که چرا دیگر از ترامپ انتقاد نمیکند: «این کار به آتش ماجرا دامن میزند.»
- نحوه برخورد با شاهزاده هری و مگان مارکل در هالیوود فاش شد
- کیم کارداشیان گفت که به دلیل هوش مصنوعی در امتحانات حقوق خود رد شده است
- مشخص شد که چرا شاهزاده هری از مگان مارکل ناراضی است
- ریچارد گیر راز یک ازدواج شاد را فاش میکند: «من یاد گرفتم که پدر خوبی باشم»
- ستاره فیلم «ایفوریا» برای اولین بار درباره رسوایی تبلیغات شلوار جین صحبت میکند: «یک واکنش غیرمنتظره»
- آلکاراز: من بیشتر اسپانیایی هستم تا یک هوادار رئال مادرید
- کیتی پری دلیل جداییاش از اورلاندو بلوم را فاش کرد
- تیموتی شالامی برای اولین بار درباره رابطهاش با جنر صحبت میکند: «دوست دارم پدر بودن را تجربه کنم.»
- شوهر سابق جنیفر لوپز این بازیگر را به خیانت متهم کرد: «مشکل از خود توست.»
- دلیل واقعی نقل مکان کیت میدلتون به خانه جدید فاش شد
- جنیفر آنیستون درباره رابطهاش با یک متخصص هیپنوتیزم صحبت میکند: «غیرعادی»
- راز انتخاب گوشوارههای کیت میدلتون در محل کار فاش شد
- شوارتزنگر طرح خود را برای «حفظ دموکراسی» در ایالات متحده مطرح کرد
- سلنا گومز در پاسخ به انتقاد هیلی بیبر گفت: «او میتواند هر چه دلش میخواهد بگوید.»
- نظر جاستین بیبر در مورد اختلاف همسرش با سلنا گومز فاش شد
- مشخص شد که چرا پسر بکهامها سعی نمیکند با والدینش آشتی کند
- نیکول کیدمن پس از انتشار خبر طلاقش، میخواهد دوباره با کیث اربن رابطه برقرار کند
- ویکتوریا بکهام درباره قلدری در مدرسه صحبت کرد: «آنها به من میگفتند احمق.»
- دمی مور درباره شغل غیرمعمولی که در ۱۴ سالگی داشت صحبت کرد
- ستاره هالیوود پس از نبردی دردناک با زوال عقل، در بستر مرگ است
- «از صمیم قلب دلم برایت تنگ شده.» لیونل مسی از ورزشگاه بازسازی شده بارسلونا بازدید کرد
- همسر جاستین بیبر در بحبوحه رسوایی گومز، از برنامه خود برای فرزند دوم خبر داد
- کیرا نایتلی توضیح داد که چرا دخترانش را از استفاده از رسانههای اجتماعی منع کرده است
- کیم کارداشیان تغییر مسیر شغلی خود را اعلام کرد
- رامی ملک، بازیگر بینالمللی متولد مصر: برای گرفتن نقشم در این فیلم جنگیدم!
- تینا کندلاکی، ۴۹ ساله، راز ظاهر آراستهاش را فاش کرد
- رونالدو گفت که حتی ترامپ هم از او محبوبیت کمتری دارد
- مشین گان کلی معتقد است که او و مگان فاکس به دلیل چشم زخم از هم جدا شدند
- نامزد سابق تیماتی عوارض تزریقات زیبایی روی بازوهایش را نشان داد: "من هرگز به آن عادت نخواهم کرد. "
- یک دلیل غیرمنتظره آنجلینا جولی را مجبور میکند وارد یک دفتر استخدام نظامی در اوکراین شود
- کندال جنر اعتراف کرد که در کودکی به خواهرش حسادت میکرده است: «حسادت میکردم.»
- شوهر خواننده سیا او را به اعتیاد به مواد مخدر متهم کرد
- همسر ۶۱ ساله باراک اوباما راز زیبایی خود را فاش کرد: «من اخم نمیکنم».
- کیم کارداشیان رابطه اش با دختر ترامپ را فاش کرد
- دلیل نفرت شاهزاده ویلیام از شاهزاده اندرو فاش شد
- کیت میدلتون و ملکه کامیلا اصرار داشتند که شاهزاده اندرو از عناوین خود محروم شود
- دلیل اینکه کیت میدلتون و پرنس ویلیام نقل مکان به خانه جدید را تسریع کردند، فاش شد
- جنیفر آنیستون در کنار نامزد هیپنوتیزم درمانگرش در سریال «عشق من» بازی میکند
- مدونا مخفیانه با معشوق ۲۹ سالهاش نامزد کرده است
- ستاره جنجالی فیلم «ایفوریا» با نامزد سابقش در ماشین مشاجره کرد
- کیت میدلتون و پرنس ویلیام به خاطر نحوه تشکر از کارکنانشان مورد انتقاد قرار گرفتهاند
- ناسا به تردیدهای کیم کارداشیان در مورد فرود بر ماه پاسخ داد
- عکس| مل گیبسون جنجالی بر سر مریم مقدس به پا کرد
- ابتلا به «ویروس اهداکننده»| یک بازیگر مشهور آمریکایی قصد دارد کلیه خود را به یک غریبه اهدا کند
- همسر سابق آرشاوین: او بنتلی من را فروخت، یک مرسدس خرید و مابهالتفاوت را برای خودش نگه داشت
- حمایت بانکها از بازار سرمایه از فردا اجرا میشود
- بورس سقوط کرده در دولت روحانی چگونه در دولت شهید رئیسی احیا شد؟
- درمان بورسی پزشکیان از زبان عبده
- جذب سرمایه ۷ همتی از بازار بورس دانشبنیانها
- وعده عناب بورسی واقعیتر شد
- چهارمین روز نزولی بازار در غیاب نیروهای صعودی
- انتخابات و دامنه نوسان مهمترین دلیل رکود فعلی بورس
- بورس با ۴ مصوبه دولت برای حمایت از بازار سهام سبزپوش میشود؟
- ۳ دلیل پایین بودن ارزش معاملات خرد در بورس
- معافیتهای مالیاتی بازار سرمایه باید استمرار پیدا کند
- بورس این روزها تحت تأثیر تداوم نرخ اخزای بالای ۳۵ درصد
- رشد شاخصهای بورس با کف سازی تکنیکال
- کاهش دامنه نوسان هم علاج بورس نزولی نبود
- سایه تامین مالی دولت بر سر بورس سنگینی میکند
- کاهش بی سابقه حجم معاملات سهام در بورسهای جهانی
- رشد شاخصهای بورس با کمک بانکیها و اعلام نرخ تسعیر ارز
- حمله به سفیر روسیه در لهستان
- معرفی سری گوشیهای Redmi K ۵۰ قبل از رونمایی رسمی
- غفوری: فکر کردن به قهرمانی از الان، سادهلوحانه است
- کاهش ۱۰ درصدی تولید هوندا در دو کارخانه
- سومین نشست دستمزد، باز هم بدون نتیجه
- لغو بلیت فروشی عمومی المپیک زمستانی چین
- گرامیداشت قربانیان هواپیمای اوکراینی در دانشگاه تورنتو
- ادامه چانهزنی چهارگانه در وین
- صرفهجویی ۱.۶ میلیارد دلاری کمیته برگزاری المپیک و پارالمپیک توکیو
- محبوبترین رشته المپیک توکیو مشخص شد
- اعلام هزینه میزبانی توکیو از المپیک و پارالمپیک ۲۰۲۰،
- آذرتاج: هیات اقتصادی جمهوری آذربایجان به ایران میرود
- بررسی عملکرد ایران در المپیک؛ جایگاه چهارمی در آسیا
- پایان وضعیت اضطراری در کشور میزبان المپیک ۲۰۲۰
- سلامت دهان و دندان و ارتباط آن با دیابت
- اسرار این دسر معروف محله شوش را فقط علیآقا میداند
- رقص خودروییها در آذر ماه!
- عامل تعیین کننده در فرآیند تمیز کردن دندانها
- تغییر فرهنگی اتفاق میافتد؟
- چرا ما روزمره به خطا میرویم؟
- من باید دیده بشم؟! واکاوی استایلهای عجیب نسل زد
- چرا این روزها گرایش به خودیاری بیشتر شده؟
- تغییر زمان... تنها رویدادی که دستهای بیگ بن را متوقف میکند
- دانشمندان کشف کردند که چرا نور سبز درد را تسکین میدهد.
- یک مطالعه جدید تایید میکند که ورزش بهترین درمان برای درد آرتروز زانو است
- چگونه شیرینیهای مناسب را انتخاب کنیم تا به فرزندتان آسیبی نرسد؟
- یک متخصص گوش و حلق و بینی در مورد درمانهای موثر گرفتگی بینی توضیحاتی ارائه داد.
- مطالعه: کاهش مصرف قند در دو سال اول زندگی فواید طولانی مدت برای قلب دارد
- راز محبوبیت محصولات کشاورزی
- اواسط زمستان، زمان تقویت سیستم ایمنی بدن است!
- ۶۰۰۰ قدم در روز خطر بیماری قلبی را در بزرگسالان مسن کاهش میدهد
- ۳ غذای برتر برای سلامت کلیهها
- علائم دهانی که ممکن است نشان دهنده کمبود ویتامین B۱۲ باشند
- چگونه در فصل سرماخوردگی و آنفولانزا بر سرفه غلبه کنیم
- به گفته دانشمندان بریتانیایی، آموزش واقعیت مجازی اشتها را کاهش میدهد.
- دانشمندان مکانیسمی را برای تقویت سلولهای بنیادی عضلانی و مقابله با ضعف عضلانی شناسایی کردند
- آیا این درست است که ساعتهای تناسب اندام به شما کمک میکنند فعالتر باشید و وزن کم کنید؟
- متخصص تغذیه: ترکیبات فرار خردل سوخت و ساز بدن را افزایش میدهد
- نوشیدنیهای شیرین خطر طاسی در مردان را افزایش میدهند، اما یک عامل خطر قویتر نیز وجود دارد
- عادات غذایی که پیری مغز را کند میکنند، شناسایی شدهاند.
- دانشمندان علت غیرمعمول سرطان را کشف کردهاند.
- محققان کشف کردهاند که چگونه زمان وعدههای غذایی میتواند بر کاهش وزن تأثیر بگذارد.
- شش دقیقه ورزش شدید، سطح یک مولکول حیاتی مغز را افزایش میدهد
- چگونه پاسخ ایمنی به استرس در افراد خوشبین و بدبین متفاوت است؟
- چرا بهتر است بدون لباس زیر بخوابیم؟
- دانشمندان دریافتهاند که برای کاهش وزن، روزانه چند قدم باید بردارید.
- سه یا چند ضربه مغزی با وخامت پیشرونده عملکرد مغز مرتبط است.
- قهوه همراه با شیر به مبارزه با التهاب دو برابر سریعتر کمک میکند.
- واکسن جدید روسی علیه سرطان پوست در اوایل سال ۲۰۲۶ به دست بیماران خواهد رسید
- دانشمندان نشان دادهاند که چگونه گجتها بر تواناییهای شناختی کودکان تأثیر میگذارند.
- دانشمندان دریافتهاند که افسردگی میتواند خطر حمله قلبی را در جوانان دو برابر کند.
- مشخص شده است که آیا ویتامین D میتواند به جلوگیری از خودکشی کمک کند یا خیر.
- یک پزشک توضیح داد که چگونه میتوان به طور مستقل سرطان را تشخیص داد.
- ارز
- طلا
- بورس
- تورم
- اخبار داغ
- ورزشی
- پربیننده
- پربحث
- یک کارشناس علوم سیاسی توضیح داد که چرا رئیس جمهور لهستان از سفر به کی یف خودداری کرد.
- بزرگترین دهانه برخورد شهاب سنگ در تاریخ زمین شناسی مدرن در چین کشف شد.
- کارشناسان: اقتصاد جهانی علیرغم افزایش تعرفههای آمریکا، در وضعیت هشدار دائمی باقی خواهد ماند
- واشنگتن در حال حرکت به سمت ایجاد مراکز کوچک برای تضمین استقلال خود در مواد معدنی حیاتی است
- پوتین یادآوری کرد که چگونه دونباس در دوران شوروی به اوکراین داده شد.
- یک راه ساده برای محافظت از خود در برابر سرطان پوست شناسایی شده است.
- مصریها به طور فزایندهای با زنان سوری ازدواج میکنند؛ آمار، رتبهبندی ازدواجها با خارجیها را نشان میدهد
- کاهش فروش گروه کالاهای لوکس فرانسوی کرینگ
- پوتین: گاهی اوقات «بیصدا» و بدون چراغها و سر و صدای کاروانهای موتوری سفر میکنم
- پوتین گفت روسیه به دنبال پایان دادن به جنگ در اوکراین است.
- دانشمندان توضیح دادهاند که چرا روند ابتلا به بیاشتهایی عصبی مشابه اسکیزوفرنی است.
- پوتین درباره خطرات هوش مصنوعی هشدار میدهد: بین ضرر کامل و استفاده بیملاحظه
- نامهای آتشین از قاهره به فیفا در مورد اثربخشی حمایت از LGBTQ+ در جام جهانی
- تفلیس از «عملیات ترکیبی» لندن علیه گرجستان خبر داد.
- نبنزیا: زلنسکی در تلاش است تا اطلاعات خود از فساد در اوکراین را پنهان کند
- آمازون به کارگیری ابزارهای هوش مصنوعی را در انبارهای خود تسریع میکند
- حکم دادگاه در مورد شریک زندگی جفری اپستین، مجرم جنسی
- زلنسکی به ترامپ در مورد انتخابات اوکراین پاسخ داد.
- غذاهایی که خطر ابتلا به سرطان روده بزرگ را افزایش میدهند، شناسایی شدند.
- اطلاعات روسیه از کشف یک معامله جدید فساد و اختلاس بین اوکراین و اروپاییها خبر داد
- پسکوف: هرگونه اقدامی که داراییهای روسیه را هدف قرار دهد، «بیپاسخ نخواهد ماند» و عواقب جدی خواهد داشت
- اروپا به انتقاد ترامپ واکنش نشان داد
- علت غیرمنتظرهای برای پیری زودرس شناسایی شد.
- حمد بن جاسم درباره پروژه راه آهن عربستان-قطر اظهار نظر میکند و آرزوهای خود را در مورد آن آشکار میسازد
- استاب گفت صلح در اوکراین از هر زمان دیگری نزدیکتر است.
- دانشمندان تهدید یک شراره خورشیدی بزرگ برای زمین را ارزیابی کردهاند.
- سناتور آمریکایی: هگزت نتوانست پاسخ روشنی در مورد برنامههای کاخ سفید در ونزوئلا ارائه دهد
- نبنزیا درباره علاقه اروپا به ادامه درگیری در اوکراین صحبت کرد.
- دانشمندان یک طوفان مغناطیسی «در مقیاس سیارهای» را روی زمین ثبت کردهاند.
- توسعهدهندگان DeepSeek در صدر فهرست «خبرسازان علمی» برای سال ۲۰۲۵ قرار دارند
- نسخهای از کتاب تاریخ فنلاند با روسیه را بخوانید! زاخارووا از «جهل» وزیر امور خارجه فنلاند انتقاد میکند
- مرتس صلح تحمیلی برای اوکراین را «غیرقابل تصور» دانست
- مشخص شده است که چرا زنان بیشتر از مردان دچار سکته مغزی میشوند.
- ترامپ: من قول کمک مالی ۲۰ میلیارد دلاری به مجارستان را ندادهام
- وزیر امور خارجه ونزوئلا: واشنگتن جنگ روانی بیسابقهای علیه ما به راه انداخته است
- اروپا نسبت به «آتشبس ارزان» و «صلح پرهزینه» برای اوکراین هشدار داده است.
- یک علت ناشناخته برای اضطراب پیدا شده است
- زلنسکی: آمریکا اوکراین را عضو ناتو نمیبیند
- زلنسکی آمادگی اوکراین را برای آتشبس در حوزه انرژی اعلام کرد.
- فواید غیرمنتظره رابطه جنسی برای سلامت روان آشکار شد
- زلنسکی اعلام کرد که اوکراین هیچ شانسی برای پیوستن به ناتو ندارد.
- دانشمندان کشف کردهاند که چرا مارها سنگ کلیه ایجاد نمیکنند.
- غرب اعلام کرده است که ترامپ «آخرین» ضربه را به زلنسکی وارد کرده است.
- دانشمندان دریافتهاند که داروی فشار خون بالا، رشد تومورهای مغزی را کند میکند.
- روسیه نزدیک به ۷۲ میلیون دلار به سازمان ملل پرداخت کرد.
- رژیم غذایی که میتواند میگرن را تسکین دهد، معرفی شد
- دومای دولتی شمارش معکوس برای پایان حکومت زلنسکی را اعلام کرد.
- یک عامل خطر برای پیشسرطان روده بزرگ در زنان جوان شناسایی شده است.
- دومای دولتی روسیه، آغازگران جنگ احتمالی بین اروپا و روسیه را معرفی کرد.
- حیوانی که تقریباً به طور کامل از مغز ساخته شده است، معرفی شد.
- آلمان انتقادهای مطرح شده در راهبرد امنیت ملی آمریکا را رد کرد
- دانشمندان راهی برای تخمین خطر ابتلا به زوال عقل ۱۰ سال قبل از ابتلا پیدا کردهاند.
- نزدیک به ۴۰۰ مدرسه در مرز با تایلند در کامبوج تعطیل شدهاند.
- جهش در یک ژن با چندین اختلال روانی مرتبط دانسته شده است.
- واشنگتن پلتفرم آنلاینی برای افشای مجرمان در میان مهاجران راهاندازی میکند
- دکلان رایس در مورد بهبود عملکردش: من بیصبرانه منتظر کمک به تیم هستم
- یک سیاستمدار اروپایی: واشنگتن در دوران ترامپ خودخواه شده و نقش خود را در جهان کنار گذاشته است
- آفریقای جنوبی درباره مشارکت خود در حل و فصل مناقشه اوکراین صحبت کرد.
- ترامپ: اروپا مجموعهای از کشورهای در حال فروپاشی است که توسط افراد ضعیف اداره میشوند
- دانشمندان کشف کردهاند که آسیب مغزی در مراحل اولیه فشار خون بالا آغاز میشود.
- مالینن از فنلاند خواست برای نجات بخش گردشگری، مرزهای خود با روسیه را باز کند
- انتخابات هندوراس: اتهامات دزدی و دخالت آمریکا در بحبوحه تعلیق شمارش آرا
- ترامپ از روی آوردن شرکای ناتو به او برای میانجیگری با اردوغان صحبت میکند
- وزیر امور خارجه مجارستان: فون در لاین بیش از هر مقام دیگری به اروپا آسیب رسانده است
- اتحادیه اروپا لهستان را از پذیرش مهاجران معاف کرد.
- تغییرات غیرمعمولی در خون افرادی که از کووید-۱۹ بهبود یافتهاند، مشاهده شده است.
- اتحادیه اروپا خواستار جلوگیری از دخالت در امور داخلی شد
- روسیه یک نانوسیال برای افزایش تولید نفت توسعه داده است.
- نماینده مجلس اوکراین: زلنسکی با تلاشهایش برای طولانی کردن مذاکرات سازش، واشنگتن را عصبانی کرد
- تراشتگن پس از مدتها به ترکیب بارسلونا بازگشت
- وزیر امور خارجه لهستان درباره جلسه اضطراری شورای دریای بالتیک صحبت کرد.
- تخمهای کروکودیل با قدمت ۵۵ میلیون سال در استرالیا کشف شدند.
- مشخص شده است که ایالات متحده در حال افزایش فشار بر اوکراین برای پذیرش طرح صلح است.
- یک عامل خطر نامشخص برای فشار خون بالا شناسایی شده است.
- واشنگتن واردات کامپیوتر و چاپگرهای چینی را به تدریج ممنوع میکند
- علاء مبارک در مورد بحران محمد صلاح اظهار نظر کرد: ستاره بودن او غیرقابل انکار است، اما رویکردش اشتباه است!
- پوتین اعلام کرد که اکثر اهداف توسعهای روسیه تا سال ۲۰۲۵ محقق خواهد شد.
- دانشمندان پیشساز غیرمنتظرهای برای بیماریهای آلزایمر و پارکینسون شناسایی کردهاند.
- مسمومیت دسته جمعی مسافران پرواز شانگهای به مسکو
- گینه بیسائو این نظریه را که رئیس جمهور سابق کودتا کرده است، رد کرده است.
- یک میزبان غیرمنتظره و مورد علاقه برای کوبی ماینو؟
- پل اسکولز فاش کرد که آرسنال برای قهرمانی در لیگ برتر باید چه چیزهایی را بهبود ببخشد
- رئال سوسیداد در حال بررسی جذب یک مهاجم است
- خیمنز، مهاجم، برنامهای برای ترک میلان در آینده نزدیک ندارد
- غایبان کلیدی، شانس پاری سن ژرمن را پیش از دیدار مقابل اتلتیک بیلبائو تهدید میکنند
- گواردیولا در مورد رقابت قهرمانی لیگ برتر: نمیخواهم مغرور به نظر برسم، اما تجربه دارم
- جایگزینی برای ابراهیما کوناته
- میکل یاورگیزار، یک تقویت کننده مورد نظر
- ویتور روکه به شایعات اشاره میکند
- گابریل ژسوس جایگزین مکس دومان در لیست لیگ قهرمانان اروپا آرسنال شد
- بکهام به اولین قهرمانی اینتر میامی در جام MLS واکنش نشان داد و گفت: «به همین دلیل مسی را آوردم.»
- ژابی آلونسو درباره وضعیت سلامتی فرانکو مستانتونو صحبت کرد
- جاکومو راسپادوری، در کانون توجه
- فابیان رویز توپ را در زمین پیاسجی رها میکند
- لیگ قهرمانان اروپا | لیورپول در میلان سلطنت میکند؛ گرینوود مارسی را به پیش میراند
- اظهارات آلونسو در آستانه بازی پیش رو مقابل سیتی در لیگ قهرمانان اروپا در پاسخ به این سوال که آیا از بازیکنان رئال مادرید ناامید شده است یا خیر
- آینده وینیسیوس روی نقل و انتقالات تابستانی رئال مادرید تأثیر میگذارد
- دیوید جیمز: لیورپول در حال حاضر بازیکنی را برای جایگزینی صلاح در نظر دارد
- نیکو گونزالس صحبت میکند
- قرارداد موریو امضا شد
- لورنزو لوکا دو خواهان در سری آ دارد
- لوئیس انریکه درباره اهمیت بازی مقابل اتلتیک بیلبائو صحبت کرد
- ناپولی در سال تقویمی ۲۰۲۵ حتی یک بازی خانگی در سری آ را نباخته است
- مشخص شد که کارواخال پس از شکست رئال مادرید مقابل سلتا به داور چه گفته است
- هانسی فلیک تغییر لامین را توضیح میدهد
- چالش بعدی ارلینگ هالند
- آنها اصرار دارند که رئال بتیس را با پابلو ماری مرتبط کنند
- لیورپول برای پنجمین بار در تاریخ و چهارمین بار در قرن بیست و یکم، اینتر را شکست داد
- فن دایک، صلاح و دیگر بازیکنان کلیدی لیورپول خواستار اخراج آرنه اسلوت هستند
- رافینیا در مورد شایعات نقل و انتقالات: «این مزخرفات را از کجا میآورند؟»
- ابهامات پیرامون لوئیس سوارز افزایش مییابد
- اخراج ژابی آلونسو را قطعی شده میدانند!
- آنها با کریستانتوس اوچه آرزوها را در هم میشکنند
- لامین یامال رکورد لیگ قهرمانان اروپا را در بین بازیکنان ۱۸ سال و کمتر به نام خود ثبت کرد
- نیمار در مورد عمل جراحی پیش رویش گفت: «من در بدترین شرایط ممکن بودم.»
- کلوپ درها را به روی رئال مادرید بست
- «کاملاً متناقض.» تلویزیون رئال مادرید از عملکرد داور در بازی مقابل سلتا انتقاد کرد
- واکنش تند پدری
- خوشبختی ژول کونده
- خولیان آلوارز، راضی؟!
- اینتر و لیورپول برای اولین بار در تاریخ خود در مرحله گروهی لیگ قهرمانان اروپا به مصاف هم رفتند
- نیمار: البته که قلب من هنوز متعلق به سانتوس است
- «یک شکست و کاری که او انجام داد، یک رسوایی است» ... شدیدترین حمله کرگر به محمد صلاح
- رونی در مورد صلاح گفت: «او کاملاً دارد میراثش را در لیورپول نابود میکند.»
- رائول به صحنه بازمیگردد... و ژوزه مورینیو سکوتش را میشکند!
- مذاکرات برای جذب پائولو دیبالا
- بهای گزافی که برای خرید ادرسون باید پرداخت شود، فاش شد
- آینده کین در بایرن مونیخ مشخص شد
- لوئیس انریکه در مورد رابطهاش با بازیکنان پاری سن ژرمن صحبت کرد
- کونده به دبل خود مقابل اینتراخت در بازی لیگ قهرمانان اروپا واکنش نشان داد
- یک تغییر اساسی در آینده اندریک؟
- سه گزینه برای خط حمله بارسلونا
- ژابی آلونسو: «بله، من حمایت تیم را احساس میکنم.»
- مایولو با ۱۰ گل زده برای پاری سن ژرمن، پس از امباپه، به سومین بازیکن جوان تاریخ این باشگاه تبدیل شد
- تاتنهام رکورد جدیدی در تاریخ لیگ قهرمانان اروپا ثبت کرد
- فلیک به پیروزی تیمش در بازی لیگ قهرمانان واکنش نشان داد
- انتقادهای خودمحور اورلین تومنی
- رئال اویدو از رئال بتیس درخواست جذب بازیکن کرده است
- کریم بنزما در آستانه امضای قرارداد است
- دکلان رایس درباره دوران کودکی ورزشکاریاش میگوید: «میخواستم در همه چیز بهترین باشم.»
- دکلان رایس در مورد استانداردهای آرسنال: «آنها از من انتظار بیشتری دارند»
- «دوش میگیرم و برمیگردم»: مصاحبهکننده محمد صلاح موضع غافلگیرکنندهای را فاش کرد
- رئال مادرید برای اخراج ژابی آلونسو شرط گذاشت
- موضع مالکان لیورپول در مورد آرن اسلات مشخص شد
- دو نامزد برای جایگزینی ژابی آلونسو در رئال مادرید مشخص شدند
- پیشنهاد وسوسه انگیز برای حیسم حسن
- دنی مورفی درباره محمد صلاح و لیورپول میگوید: «او دارد مشکل ایجاد میکند.»
- هدف رئال مادرید برای خط هافبک مشخص شد
- دکلان رایس در مورد رقابت در آرسنال میگوید: «همه میخواهند شروع کنند.»
- برنامه بازیهای مرحله یکهشتم نهایی کوپا دل ری مشخص شد
- باشگاه پاری سن ژرمن به شایعات جدایی ماتوی سافونوف واکنش نشان داد
- رئال مادرید در مورد زین الدین زیدان تصمیم گیری میکند
- مدافع بارسلونا در مورد دیدار پیش رو مقابل اینتراخت در لیگ قهرمانان اروپا صحبت کرد
- تصمیم «آسانی» که آلخاندرو گارناچو گرفت
- در صورت لغو محرومیت دوپینگ، مودریک میتواند دوران حرفهای خود را در فرانسه یا اسپانیا ادامه دهد
- تصمیم کلیدی توسط آنتوان سمنیو
- فوت مرکاتو: باشگاه عربستانی آماده پرداخت ۱۰۰ میلیون یورو برای میسون گرینوود است
- یازده باشگاه راه یان دیوماند را دنبال میکنند!
- لوئیس سوارز برنامهای برای تمدید قرارداد با اینتر میامی ندارد
- سرژ گنابری آیندهاش را مشخص کرد